RÉSISTANCE CLINIQUE ET RÉSISTANCE VIROLOGIQUE
La résistance clinique se définit par l’absence d’amélioration des symptômes de l’infection malgré l’existence d’un traitement bien conduit et à dose efficace, par exemple la persistance de symptômes digestifs pour une colite à CMV (cytomégalovirus) traité par ganciclovir. Elle peut aussi être suspectée lors de l’apparition de symptômes chez une personne sous prophylaxie antivirale, par exemple l’apparition de lésions génitales d’HSV-2 sous valaciclovir.
La résistance virologique se définit par la persistance d’une réplication virale malgré l’existence d’un traitement bien conduit et à dose efficace ou l’apparition d’une réplication virale chez une personne sous prophylaxie antivirale. La mise en évidence de la résistance virologique nécessite la quantification des génomes viraux dans le sang (ex. mesure de la charge virale sanguine du HIV [virus de l’immunodéficience humaine], HCV [virus de l’hépatite C], HBV [virus de l’hépatite B], CMV…), dans les liquides biologiques (ex. quantification du HSV [virus de l’herpès simplex], CMV, VZV (virus de la varicelle et du zona) dans le liquide céphalorachidien, l’humeur aqueuse ou le liquide bronchoalvéolaire…) ou dans les tissus (ex. quantification du CMV dans une biospie digestive…).
Le suivi de la charge virale sous traitement a un rôle très important. Il permet de valider l’efficacité des traitements antiviraux avec la mise en évidence d’une baisse de la réplication virale. De manière générale, la réplication virale doit être inhibée le plus rapidement possible (diminution de la charge virale d’un log10 par semaine) et jusqu’à la disparition complète du virus (absence de détection du virus par PCR [Polymerase Chain Reaction]). Ceci permet de minimiser le risque de générer des virus mutants et donc de prévenir l’émergence de virus résistants.
DÉTERMINATION DE LA RÉSISTANCE
Comme nous l’avons précisé précédemment, trois éléments majeurs interviennent dans la résistance aux antiviraux, le virus, le patient et l’antiviral lui-même.
Les sous-dosages en antiviral (par manque d’observance du traitement, posologies inadaptées ou interactions médicamenteuses) peuvent être objectivés par des dosages plasmatiques des antiviraux. Ces sous-dosages peuvent (en laissant répliquer les virus les moins sensibles) être à l’origine de la sélection de souches résistantes. La réponse immunitaire du patient peut être explorée par l’évaluation de la réponse immunitaire humorale contre le virus (sérologie virale) ou cellulaire (test IGRA [Interferon Gamma Release Assays] comme le quantiféron ou l’ELISPOT). Enfin la résistance du virus aux antiviraux peut être mise évidence par des tests phénotypiques et/ou génotypiques. Seules ces analyses seront développées ci-dessous.
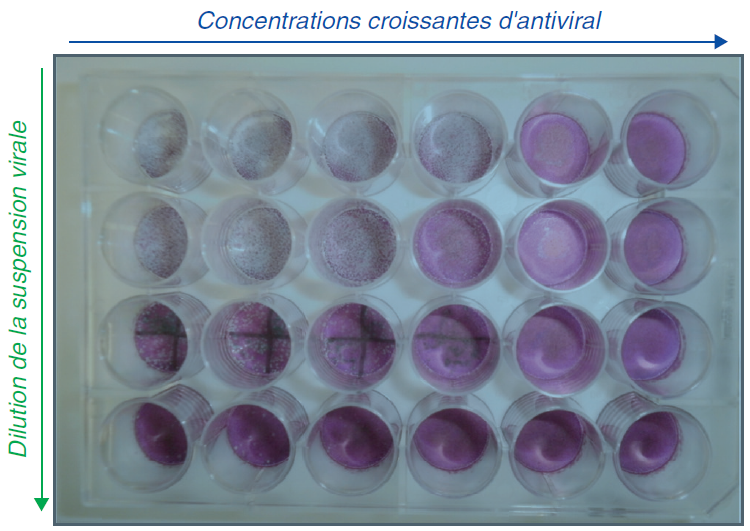
DÉTERMINATION PHÉNOTYPIQUE DE LA RÉSISTANCE DES VIRUS AUX ANTIVIRAUX
Comme nous l’avons précisé précédemment, trois éléments majeurs interviennent dans la résistance aux antiviraux, le virus, le patient et l’antiviral lui-même.
Les sous-dosages en antiviral (par manque d’observance du traitement, posologies inadaptées ou interactions médicamenteuses) peuvent être objectivés par des dosages plasmatiques des antiviraux. Ces sous-dosages peuvent (en laissant répliquer les virus les moins sensibles) être à l’origine de la sélection de souches résistantes. La réponse immunitaire du patient peut être explorée par l’évaluation de la réponse immunitaire humorale contre le virus (sérologie virale) ou cellulaire (test IGRA [Interferon Gamma Release Assays] comme le quantiféron ou l’ELISPOT). Enfin la résistance du virus aux antiviraux peut être mise évidence par des tests phénotypiques et/ou génotypiques. Seules ces analyses seront développées ci-dessous.

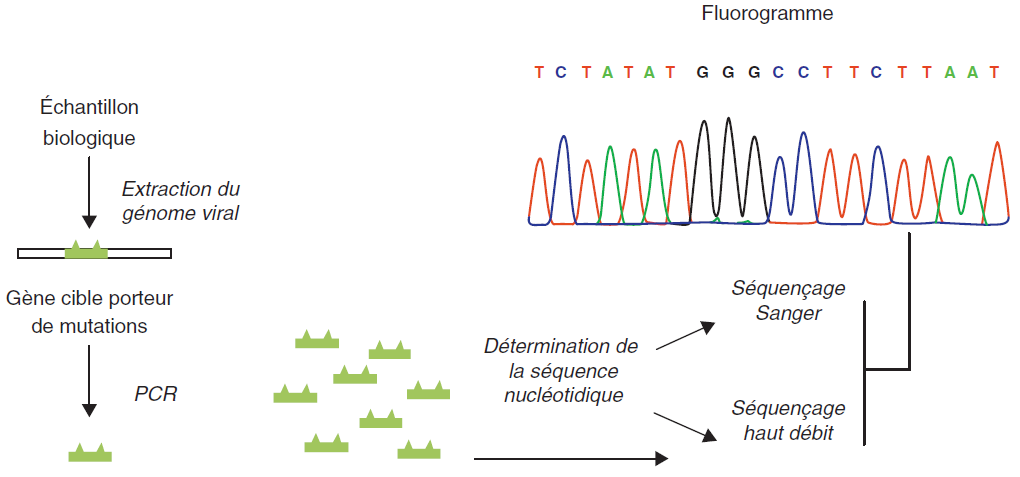
DÉTERMINATION GÉNOTYPIQUE DE LA RÉSISTANCE AUX ANTIVIRAUX
Les tests de résistance génotypiques peuvent être réalisés sur des isolats viraux mais aussi directement sur les prélèvements. Ils reposent sur la recherche de mutations sur les gènes viraux cibles des antiviraux et associés à la résistance. Il est nécessaire de différencier les mutations associées à la résistance de celles liées au polymorphisme viral (mutations sans effet sur la sensibilité aux antiviraux). Les tests sont réalisés par les laboratoires des CHU pour certains virus, entre autres HIV, HBV, HCV, CMV, HSV ou VZV*. Ils présentent un intérêt majeur pour le suivi des infections chroniques actives traitées au long cours. Ainsi, l’émergence de virus résistants détectée précocement permet l’optimisation de l’utilisation des antiviraux. Ces tests ont une valeur diagnostique avant l’initiation d’un traitement ou devant un échec thérapeutique, et une valeur pronostique pour anticiper une réponse thérapeutique.
La technique consiste à amplifier (PCR) puis séquencer les gènes codant les protéines cibles (enzymes virales, protéines de liaison au récepteur/corécepteur) des antiviraux à étudier. Le séquençage repose classiquement sur la technique de Sanger, considérée à ce jour comme la technique de référence. La comparaison de la séquence en acides aminés obtenue avec celle d’une souche sensible (« sauvage ») permet d’identifier les mutations connues comme associées aux résistances (figure IV.3.3). Des algorithmes, mis à jour périodiquement en fonction de l’évolution des connaissances, permettent d’interpréter des profils de mutations pour chaque antiviral en termes de sensibilité et de résistance. Le séquençage classique a une sensibilité de 20 %, c’est-à-dire qu’il ne détecte une mutation que lorsqu’au moins 20 % de la population virale la possède. Les techniques de séquençage haut débit ou de nouvelle génération (NGS) permettant de détecter des variants de faible fréquence sont beaucoup plus sensibles, mais elles nécessitent de déterminer des seuils cliniques pertinents afin de pouvoir prédire l’impact clinique de chaque mutation sur la réponse thérapeutique du patient.
CONCLUSION
La prise en compte de la résistance consiste à choisir l’antiviral ou les antiviraux les plus efficaces aux posologies usuelles sur les populations virales détectées, les mieux tolérés et enfin de prise facile, assurant une adhésion/observance au choix du patient au traitement antiviral sur le long terme. En pratique, en cas de résistance confirmée, l’adaptation thérapeutique est discutée dans des réunions de concertation pluridisciplinaire (cliniciens, virologues, pharmacologues).
| Points clés La résistance aux antiviraux s’explique par l’existence de variants minoritaires porteurs de mutations au sein de la quasi-espèce virale qui peuvent être sélectionnés sous la pression du traitement antiviral. Les tests de résistance phénotypiques nécessitent l’isolement préalable du virus en culture cellulaire. Les tests de résistance phénotypiques sont réalisés dans les centres nationaux de référence. Les tests de résistance génotypiques reposent sur des techniques de séquençage et sont réalisés dans les laboratoires des CHU. Les tests de résistance génotypiques nécessitent des bases de données qui doivent être mises à jour régulièrement. Pour les tests de résistance génotypiques, le séquençage par la technique de Sanger reste la technique de référence mais le séquençage NGS prend une place de plus en plus importante. |
